AutoDock Vina
AutoDock Vina
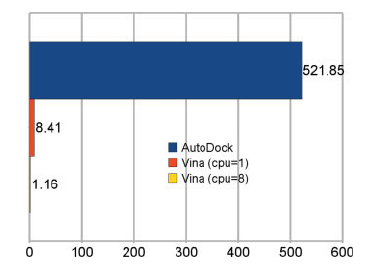
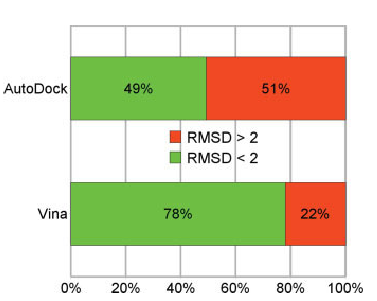
Program wykorzytywany do dokowania molekulanego oraz screeningu układów białko-ligand. Implementacja nowych algorytmów optymalizacji, funkcji dopasowania (scoring function) oraz wprowadzenie możliwości zrównoleglenia obliczeń zaowocowało wynikami lepszej jakości uzyskanymi w do 2 rzędów krótszym czasie względem poprzedniej generacji oprogramowania (AutoDock4.x).
[ilustracje]
Przypływ danych - w każdym etapie można zastosować alternatywne oprogramowanie.
Przygotowanie pliku wsadowego w MGLTools (*.PDBQT).
- przygotowanie struktury białka (dodanie wodorów, określenie rozmiaru przestrzeni dopasowania)
- przygotowanie ligandu (określenie wiązań rotacynych)
Przeprowadzenie obliczeń w AutoDock Vina.
- AutoDock Vina uruchamiamy skryptem sub-autodock-vina
Interpretacja wyników w PyMOL.
Szczegóły na stronie projektu http://vina.scripps.edu/ oraz w : "O. Trott, A. J. Olson, AutoDock Vina: improving the speed and accuracy of docking with a new scoring function, efficient optimization and multithreading, Journal of Computational Chemistry 31 (2010) 455-461"
W publikacjach, w których do liczenia korzystano z AutoDock Vina, autor prosi cytować ww. artykuł. \* Za"O. Trott, A. J. Olson, AutoDock Vina: improving the speed and accuracy of docking with a new scoring function, efficient optimization and multithreading, Journal of Computational Chemistry 31 (2010) 455-461"